
[Vol.8 No.4] DNA 저장기술의 과거, 현재, 그리고 미래
작성자
관리자작성일자
2018-12-13 10:36조회수
429
DNA 저장기술의 과거, 현재, 그리고 미래
현재 사회관계망 (SNS) 서비스, 클라우드 저장서비스, 디지털 영상장치, 사물인터넷 (IoT) 등 다양한 데이터 생성원에 의해 데이터 양은 매우 급격한 속도로 팽창하고 있다. 보도에 따르면 2017년 한 해동안 데이터센터를 건립하는데 미국에서만 2조원 이상의 비용이 소요되었으며, 그 비용은 매년 2배씩 증가하는 경향을 보이고 있다. Cisco는 2019년까지 IoT 데이터가 500ZB에 이를 것으로 예상하고 있다. 생산한 데이터들을 모두 저장한다고 했을 때 현재 데이터센터의 구성 라인업인 하드디스크드라이브 (HDD), 솔리드스테이트드라이브 (SSD), 자기테이프로는 조만간 그 수요를 감당할 수 없을 것으로 예상된다. 발생하는 데이터 중 많은 부분은 오랜 기간동안 엑세스되지 않을 가능성이 크다. 이러한 콜드데이터는 현재로서 자기테이프에 저장되게 되는데, 비교적 저장밀도가 높지만 수명이 10~15년 밖에 되지 않고 임의접근 (random access)가 불가능하다는 단점이 있다.
2012년 Church 그룹에 의해 DNA가 저장장치로서 사용될 수 있다는 가능성을 보인 이래, DNA 저장장치는 기존 저장장치를 대체할 수 있는 유력한 후보로 각광받고 있다. DNA 저장장치는 기존 대비 1000배 이상의 초고밀도를 가지고, 100년 이상의 수명이 가능하며, 전력을 거의 소모하지 않는 장점을 보인다. 이러한 점에 주목하여 Microsoft Research는 2020년까지 DNA 저장장치를 이용하여 데이터센터를 건립하기로 발표하고, University of Washington과 공동연구를 진행하여 DNA 저장장치 연구 및 상용화에 앞장서고 있다. 또한 CATALOG Technology는 보스턴 지역에 기반한 스타트업 회사로 2019년에 DNA 저장서비스를 상용화하는 것을 목표하고 있다.
DNA에 데이터를 쓰고 읽기 위해서는 부호화, 합성, 저장, 분리, 시퀀싱, 복호의 과정이 필요하다. 부호화 및 복호는 데이터를 쓰고 읽는 과정에서 발생하는 오류를 제어하여 가장 큰 저장용량을 달성하기 위해 필요하다. 저장하고자 하는 데이터는 부호화를 거치고 난 후 네 가지 염기인 A, G, C, T에 해당하는 4진 알파벳의 시퀀스로 변환되는데, 이 시퀀스에 따라 올리고 (oligo)라 불리는 짧은 DNA를 합성하게 된다. 긴 길이의 DNA를 안정적으로 합성하고 시퀀싱하는 것은 아직 기술적으로 쉽지 않으므로 보통 200~1000 이내의 길이의 올리고들을 이용하고, 이에 따라 데이터를 블록단위로 분할하여 부호화를 수행한다. 합성된 올리고들은 일반적으로는 액체타입의 올리고 풀 (pool)을 형성하여 튜브나 플레이트 형태에 담겨서 저장된다. 시퀀싱을 위해서는 여러 가지의 전처리 절차를 거치게 되는데, PCR (polymerase chain reaction)이라고 불리는 개체 증식 절차를 거쳐서 시퀀싱이 원활하게 수행될 수 있도록 한다. 현재의 시퀀싱 플래폼들은 무작위 추출을 통해서 시퀀싱을 하게 되므로 각 올리고에는 고유의 인덱스가 있어야만 읽은 데이터의 순서를 알 수 있으며 복호가 가능하다.
DNA 저장장치의 연구는 2012년으로 거슬러 올라간다. Havard 대학의 George M. Church 연구그룹은 0.65MB의 데이터를 DNA에 저장하는데 성공하고 Science에 논문을 게재했다. 이후로 여러 연구그룹들에 의해 DNA에 데이터를 저장하는 방법들이 시도되었다. 이 때까지는 주로 오류를 제어하기 위해서 반복되는 데이터들을 주로 이용하거나 간단한 대수적 구조의 부호를 사용하였다. 그러다가 2017년부터는 DNA 저장방법에 다양한 breakthrough들이 일어나게 되는데, Erlich와 Zielinski는 파운틴 부호를 이용하여 2.11MB의 데이터를 저장하는데 성공하고 Science에 논문을 게재하였고, Yadzi는 처음으로 nanopore 시퀀싱 플랫폼을 이용하여 데이터를 저장하는데 성공하여 Scientific Report에 논문을 게재한다. 2018년 U. Washington의 Organick은 200.2MB의 대용량 데이터를 임의 접근이 가능하도록 저장하는데 성공하여 Nature Biotechnology에 논문을 게재한다.
현재 DNA 저장장치의 상용화에 있어서 가장 문제가 되는 부분은 합성의 비용 및 속도이다. 물론 합성 및 시퀀싱 비용 모두 시간이 지남에 따라 기하급수적으로 감소하고는 있지만, 시퀀싱 비용은 비교적 저렴한 반면, 합성의 경우 현재 일반적인 방법으로는 1MB를 저장하는데 1천만원에 가까운 비용이 들어가고 있다. CATALOG Technology의 경우 이러한 문제점을 해결하고자 합성을 빠르게 할 수 있는 기술을 통해서 상용화를 시도하고 있으며, 하루에 1Tbit를 합성할 수 있는 속도를 달성하는 것으로 알려져 있다. 한편 DNA 저장장치를 위한 오류제어 기술은 최적의 기술이 알려지지 않은 가운데 초기 단계의 연구들이 진행되었다고 볼 수 있다. 상용화까지 가기 위해서는 보다 현실적인 조건에서 최적의 오류제어 기법이 연구되어야 할 것으로 보인다. NAND 플레시 메모리의 성공에 고성능 오류정정부호를 적용한 컨트롤러가 큰 기여를 했듯이 DNA 저장장치의 성공에도 최적화된 오류제어 기술이 큰 역할을 하게 될 것으로 전망된다.
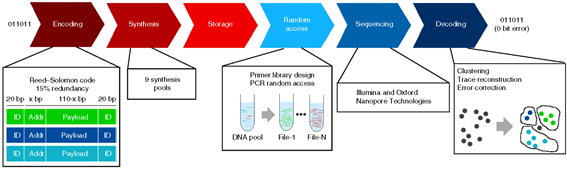
(출처: L. Organick et al., “Random access in large-scale DNA data storage,” Nature Biotechnology, 2018.)
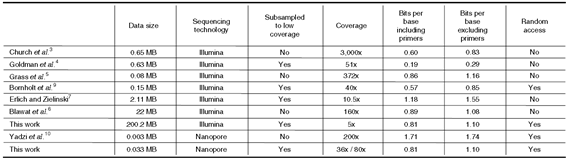
(출처: L. Organick et al., “Random access in large-scale DNA data storage,” Nature Biotechnology, 2018.)
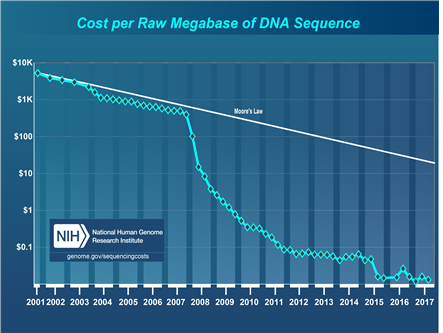
(출처: National Human Genome Research Institute, genome.gov/sequencingcosts)